单倍型基因组组装工具 --- Gamete-binning
Background
Gamete binning 是一种基于单倍体配子的单细胞测序的基因组组装方法,能够将全基因组测序 reads 分离成单倍体特异性 reads,在组装每个单倍体的 reads 后,使用源自配子的遗传图谱将 contigs 搭建到染色体水平。为了获得单倍型基因组组装,一种常见的策略是近交或产生双单倍体基因型以实现纯合基因组的组装。
chromosome sorting:染色体分选,在测序前通过实验手段分离单个染色体,从而能够对单个单倍型进行测序和组装;但这依赖于荧光强度或光散射来区分特定的染色体。在很多时候,并不能很好的将每一条染色体区分开。
Strand-seq :这是一种单细胞技术,不需要亲本或配子,可用于使用遗传图谱技术根据染色体、单倍型定相和 scaffold 对 长 reads 进行聚类;然而,生成 Strand-seq 数据的困难限制了其在少数模型物种中的应用。
high-throughput chromosome conformation capture (Hi-C):染色体构象分析,包括能够以前所未有的规模检测染色质相互作用的 Hi-C 技术,已成功应用于多种物种的单倍型定相和基因组 scaffold。
trio binning 基于在组装之前根据亲本基因组之间的基因组差异将全基因组测序 reads 分离成单倍型特异性 reads 集。但这局限于该物种父母基因组可用,若需要组装一种新物种的单倍型,则这种策略的单倍型定相方法就不适用了。
获得染色体水平的单倍型基因组,有助于我们理解基因组差异的全部复杂性(包括各种结构重排)。遗传图谱可以可靠的帮助解决错误组装并知道染色体水平的 scaffolds 组装;然而,遗传图谱的生成依赖于大量的减数分裂重组,这通常意味着对数百个重组基因组进行基因分型。
Introduction
Gamete binning:可生成染色体水平、单倍型分辨的基因组组装方法;且独立于亲本基因组或重组后代。
Gamete binning 首先从目标个体分离出配子核,然后对数百个单倍体配子基因组进行高通量单细胞测序。配子基因组中序列变异的分离能够直接将所有变体定相为两个单倍型,而后用于将全基因组测序 reads 进行遗传作图和分选成不同的 read sets — 每个 read sets 代表不同的单倍型。组装这些独立的 read sets 便可以得到单倍型解析的基因组组装,同时可以使用配子基因组衍生的遗传图谱将其搭建到染色体水平。
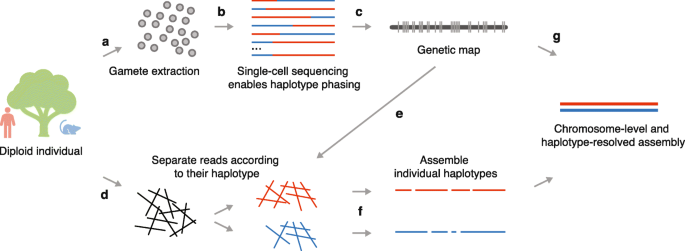
Gamete binning组装流程a、提取配子核
b、单倍体配子的单细胞基因组测序和单倍型定相
c、基于配子基因组中的重组模式构建遗传图谱
d、目标物种体细胞材料的
long reads测序e、基于遗传连锁群使用定相好的等位基因分离
long readsf、每个连锁群的每个单倍型的独立组装
g、使用配子衍生的遗传图谱将
scaffold组装到染色体水平
References
[1] Campoy, J.A., Sun, H., Goel, M. et al. Gamete binning: chromosome-level and haplotype-resolved genome assembly enabled by high-throughput single-cell sequencing of gamete genomes. Genome Biol 21, 306 (2020). https://doi.org/10.1186/s13059-020-02235-5