Dot Plot 绘制
点阵图通常用于直观地比较两组序列,可视化呈现碱基的插入、删除、反转或者重复。它们可以使用可变的线条粗细、线条形状或者颜色来表示相似性差异。
前言
现有的点阵图绘制工具可分为两代。
一种是 Unix 命令行工具生成静态图形。其中包括 tupple_plot 和 dot-matrix 它们通常将两个处理步骤链接在一起,第一步生成的文件将作为第二步的输入文件,从而生成图像。其致命缺点是 仅限于单条序列 (fasta) ,且不能与建立的图像进行交互。
第二代软件包则是采用 java 开发,以便独立于平台与用户交互。包括 JDotter 、Gepard 和 r2cat 。这一代 Dot Plot 绘制工具允许用户进行交互,对用户来说这是相当友好的。可以根据自己的需求对生成的图像进一步修饰。
新一代的点阵图可视化工具是基于 JavaScript,不过用户在上传坐标和索引文件之前必须先生成坐标和索引文件,才能渲染点阵图。早期的这一代工具:https://dnanexus.github.io/dot/
D-GENIES
今天的主角是 D-GENIES,一个交互式、快速且易于使用的独立的 Web应用程序。D-GENIES 能够在 1h 10 min 内生成完整的人类与黑猩猩基因组点阵图。
其中序列比对程序采用的是 minimap2 ,适合大型基因组的序列比对。
D-GENIES 适配 Unix 和 Windows;当然也有网页端的。详情请移步:D-GENIES - Dotplot large Genomes in an Interactive, Efficient and Simple way (inrae.fr)
这里我们尝试一下网页端的 D-GENIES:
页面十分简洁,允许上传的文件格式有:.fa .fasta .fna .fa.gz .fasta.gz .fna.gz
填写了邮箱,任务完成之后便会收到邮件通知,还是很方便的,且支持上传的 fasta 文件高达 3Gb。而对于人类基因组或其他大型基因组的 Dot Plot 可以选择本地的 D-GENIES。
Submit 之后就能看到序列文件在缓慢上传中(这将取决于你的网速)
结果解读
强烈建议将序列文件进行压缩之后再上传,因为 D-GENIES 服务器在国外,网速还是相对慢的,压缩文件可以为我们节省很多时间。文件上传之后,到出结果是很快的,几百兆的序列在十几分钟之内就能将点阵图绘制成功,所以我们没必要将时间浪费在文件上传上。
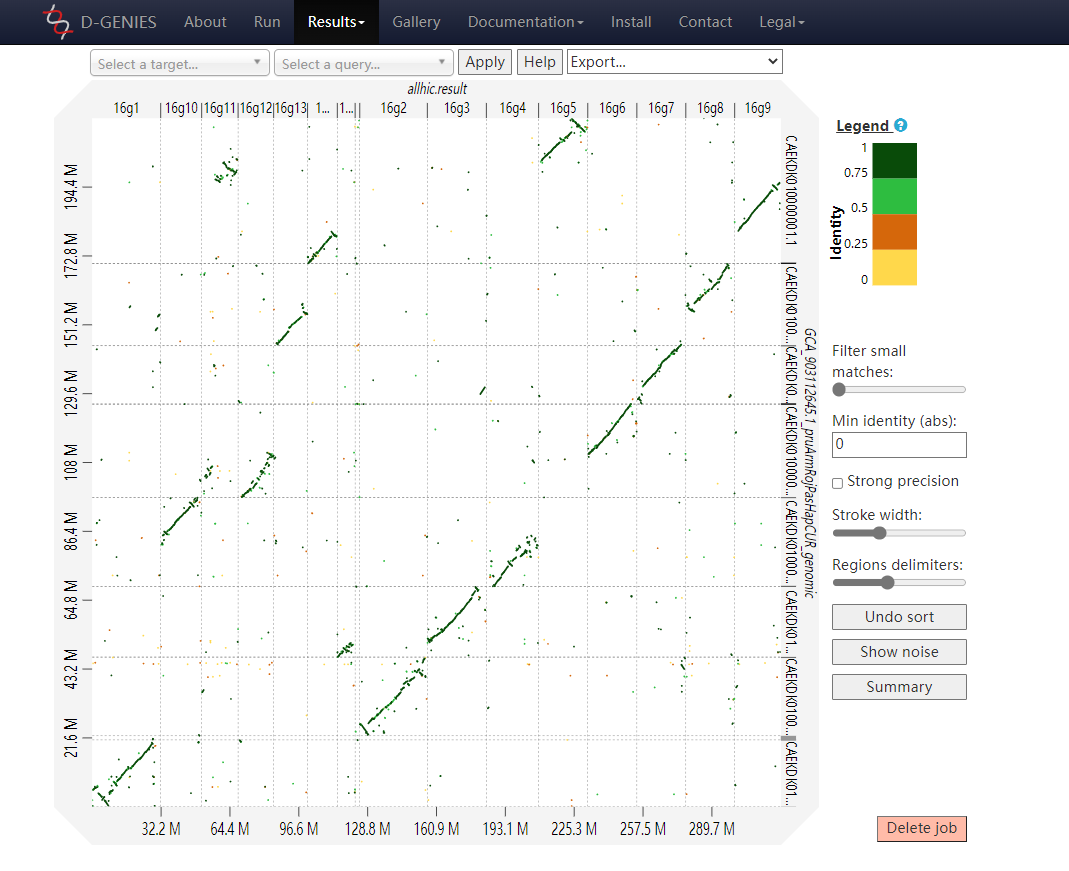
上图与上面上传的文件无关(我是另外找的两个序列)。
D-GENIES 运行还是很快的,网络稳定的情况下,200 Mb 基因组在十分钟内就能绘制出 Dot-Plot。接下来我们来看看上面的点阵图。上图便是 D-GENIES 的结果页面,你还可以进行 Strong precision — 只显示精度高的点,即隐藏相似度低于 75% 的点;Sort contigs — 对 contigs 进行排序;Hide noise — 隐藏噪点;Summary — 统计整体的 identity 分布情况。
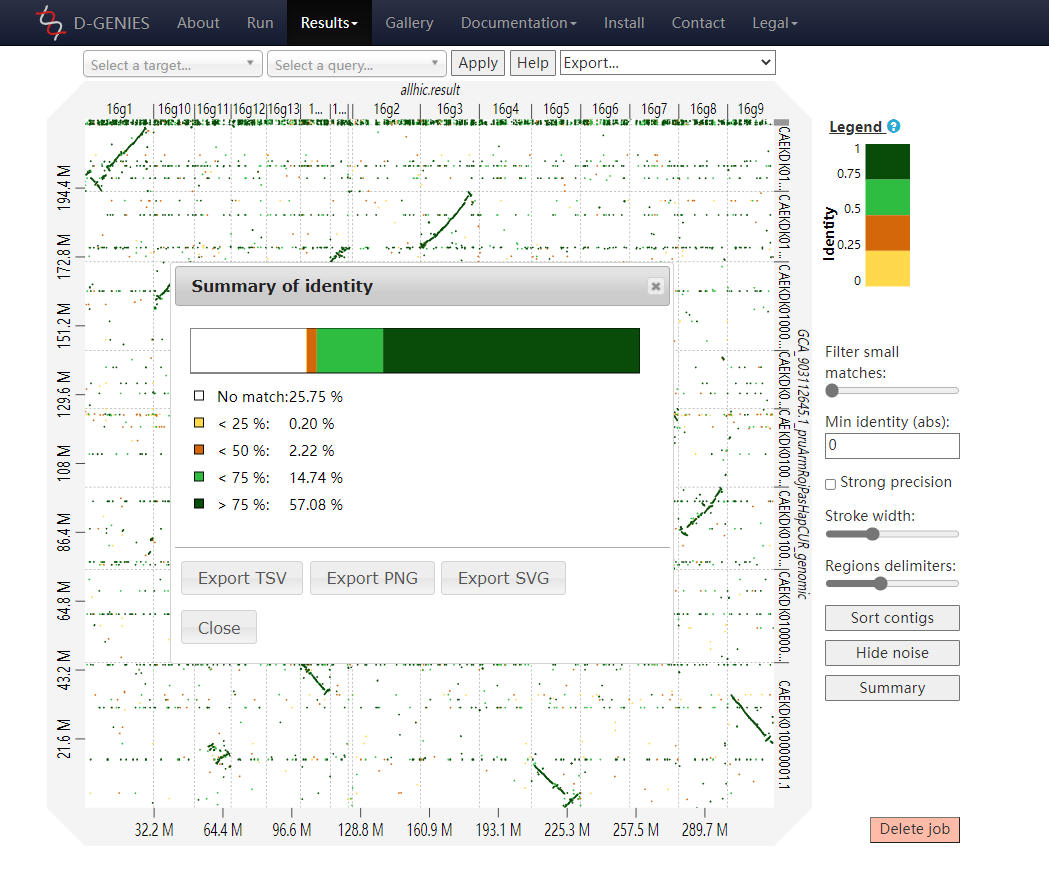
此外,还可以将生成的点阵图保存各种格式,如 png、svg 等。
Dot Plot 解读
这一部分将记录 Dot-Plot 能传递的信息有哪些,参考链接:D-GENIES - Dotplot large Genomes in an Interactive, Efficient and Simple way (inra.fr)
点阵图是一种递归图,在生物信息学中,可以比较两个生物序列之间并识别它们之间的相似性区域。
Match (匹配)
当两个生物序列相似度很高时,它们被认为是相同的,即两者是 Match。
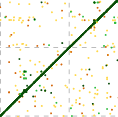
Gap(空位)
点图可用于检测两个样本之间的间隙:仅存在于一个样本中、两个匹配区域之间的小序列。
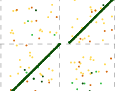
Inversion(倒置)
两个样本中存在但顺序不同的序列。

Repeats(重复)
点图可用于检测重复区域:在样本中重复多次的序列。

参考文献
[2] D-GENIES - Dotplot large Genomes in an Interactive, Efficient and Simple way (inra.fr)
[3] D-GENIES - Dotplot large Genomes in an Interactive, Efficient and Simple way (inra.fr)